German Research Foundation continues to fund the CRC 1182 at Kiel University in a third funding phase
Four more years of metaorganism research in Kiel
Upcoming Events
Recent News
Recent Publications
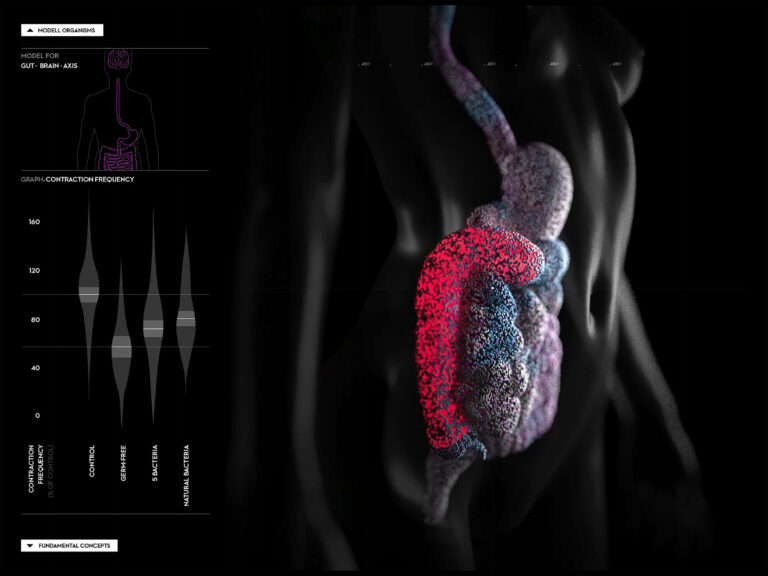
Metabolic model predictions enable targeted microbiome manipulation through precision prebiotics
Georgios Marinos, Inga K. Hamerich, Reena Debray, Nancy Obeng, Carola Petersen, Jan Taubenheim, Johannes Zimmermann, Dana Blackburn, Buck S. Samuel, Katja Dierking, Andre Franke, Matthias Laudes, Silvio Waschina, Hinrich Schulenburg, Christoph Kaleta (2024): Metabolic model predictions enable targeted microbiome manipulation through precision prebiotics. Microbiology Spectrum February 2024 Volume 12 Issue 2 e01144-23 DOI: 10.1128/spectrum.01144-23
Developing science outreach events based on stakeholders’ objectives and expectations – A case study of a lecture day for schools
Claussen C, Enzingmüller C, Kremer K, Schulenburg H, Parchmann I (2023) Developing science outreach events based on stakeholders’ objectives and expectations – A case study of a lecture day for schools. Sciendo 6:49–66. doi: 10.23770/ristal-2023-3
Communicating the Metaorganism – PR Project within the CRC 1182
The CRC 1182 Community
Collaborative Research Centre 1182
From protists to humans, all animals and plants are inhabited by microbial organisms. There is an increasing appreciation that these resident microbes influence fitness of their plant and animal hosts, ultimately forming a metaorganism consisting of a uni- or multicellular host and a community of associated microorganisms.

Speaker
Prof. Dr. Dr. h.c. Thomas C.G. Bosch
Kiel University
Zoological Institute
Am Botanischen Garten 1-9
D-24118 Kiel

Vice-Speaker
Prof. Dr. Hinrich Schulenburg
Kiel University
Zoological Institute
Am Botanischen Garten 1-9
D-24118 Kiel
+49 431 880 4143
+49 431 880 2403
hschulenburg@zoologie.uni-kiel.de